2024.01.26
【プレスリリース】分子シミュレーション×AIでトランスポータータンパク質の未解明構造を明らかにする
【発表のポイント】
- トランスポータータンパク質は、自身の構造を切り替えることで特定の分子を細胞内外へ輸送する
- 分子シミュレーションと構造予測AIであるAlphaFold2によって、シュウ酸トランスポーターについて未解明であった内開き構造とその構造変化を促進する重要アミノ酸残基の予測に成功した
- 分子シミュレーションと構造予測AIを組み合わせた今回のアプローチは、今後様々なトランスポータータンパク質の未解明構造研究への応用が期待できる
【概要】
自然科学研究機構・分子科学研究所の大貫隼助教、Jaunet-Lahary特任研究員、岡崎圭一准教授と岡山大学学術研究院医歯薬学域(薬)の山下敦子教授の研究グループは、腸内のシュウ酸分解菌がもつトランスポータータンパク質の未解明構造とその重要アミノ酸残基を、分子動力学シミュレーションと構造予測AIのAlphaFold2を用いて明らかにしました。 本研究成果は、国際学術誌 The Journal of Physical Chemistry Letters に、2024年1月12日付でオンライン掲載されました。
1. 研究の背景
シュウ酸は野菜やナッツ類などに豊富に含まれ、日々の食事を通して我々の体内に摂取されます。体内のシュウ酸濃度が過剰になるとカルシウムと結合して、尿路結石の原因となってしまいます。しかし我々の腸内にはシュウ酸を吸収して分解する細菌が共生していて、シュウ酸濃度の増加を抑えてくれています。腸からのシュウ酸の吸収は、この腸内細菌の持つシュウ酸トランスポーターが担っています。
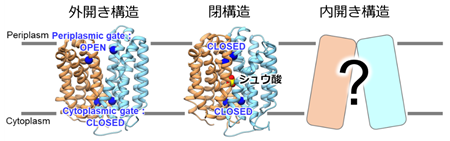
トランスポータータンパク質※1は、シュウ酸トランスポーター以外にも数多くの種類が存在しそれぞれ特定の分子を輸送しますが、いずれも交互アクセスメカニズムという仕組みで輸送物を運んでいると考えられています(図1)。この交互アクセスメカニズムにより、シュウ酸トランスポーターは、まず外開き構造をとり腸内のシュウ酸を取り込み、閉構造を経由して、内開き構造に変化して細菌内部にシュウ酸を運びます。その後シュウ酸の分解物(ギ酸)を内開き構造で取り込み、逆の経路を辿って最終的に外開き構造となって腸へ排出します。
シュウ酸トランスポーターが輸送する仕組みを理解するためには、交互アクセスメカニズムの各構造を知ることが不可欠です。我々のグループでは、これまでに外開き構造と閉構造を明らかにしました。しかし唯一、内開き構造は従来手法により実験的に解析することが困難であり、シュウ酸トランスポーターの理解を阻んでいました。そこで本研究では、分子動力学シミュレーション※2、さらに高精度なタンパク質構造予測AIとして近年注目されているAlphaFold2※3を用いて、シュウ酸トランスポーターの内開き構造の解明を目指しました。
2. 研究の成果
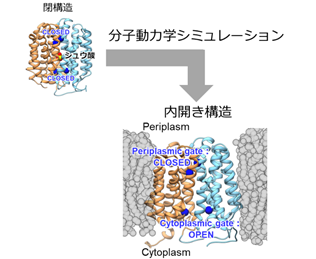
未解明の内開き構造を明らかにするため、まず既知の閉構造を出発点に分子動力学シミュレーションを行い、閉構造から内開き構造への変化を促進するため、トランスポーターの相互作用を人為的に弱める手法(Gaussian accelerated分子動力学法)を駆使することで、期待通り内開き構造を得ることに成功しました(図2)。
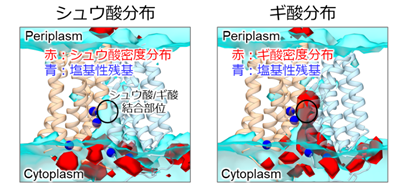
つづいて、シュウ酸、ギ酸が内開き構造へ結合するか分子動力学シミュレーションにより観察した結果、ギ酸のみが内開き構造の内部へ取り込まれることが明らかになりました(図3)。シュウ酸分解菌は、シュウ酸を吸収した後、ギ酸を取り込んで腸内へと排出するので、トランスポーターの構造自体にシュウ酸とギ酸を選別する仕組みが備わっていることは理にかなっているといえます。
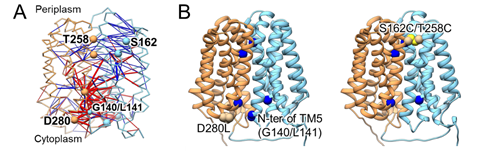
閉構造から内開き構造への変化の際、トランスポーターを構成するアミノ酸残基の間の結合にいくつか顕著な変化(結合形成と切断)が見られました(図4A)。こうしたアミノ酸残基は内開き構造への変化に重要な役割をもっていると期待されます。このことを検証するため、次に高精度なタンパク質構造予測AIであるAlphaFold2を用いました。AlphaFold2はアミノ酸配列を入力として高精度な構造予測が可能であることから近年注目されていますが、一方で、トランスポーターのように複数の構造をとるタンパク質では、特定の構造ばかり予測してしまう問題点も報告されています。シュウ酸トランスポーターでも、野生型アミノ酸配列に対しAlphaFold2は既知の外開き構造しか予測しませんでした。しかし、ここで分子シミュレーションによって特定したアミノ酸残基に変異を導入すると、内開き構造も予測できるようになりました(図4B)。つまり、今回の変異箇所は確かに内開き構造への変化の鍵となる重要アミノ酸残基であり、シュウ酸トランスポーターの構造変化を促す変異候補を特定できました。
3. 今後の展開・この研究の社会的意義
本研究では、分子動力学シミュレーションと構造予測AIを組み合わせることで、タンパク質の未解明構造およびその構造変化に重要なアミノ酸残基を特定しました。この手法を今回はシュウ酸トランスポーターに適用しましたが、他のトランスポーターにも活用できる可能性があります。トランスポーターの中には、輸送の異常が我々の疾患に関与するものもあり、今回の計算科学アプローチは、こうした疾患の理解と治療法開発の基礎となるトランスポーター研究を促進すると期待されます。
4. 用語解説
- ※1 トランスポータータンパク質
細胞膜に存在する膜タンパク質であり、外開き構造と内開き構造を交互に変化すること(交互アクセスメカニズム)によって特の分子を細胞内外へ輸送する - ※2 分子動力学シミュレーション
タンパク質分子や周囲の水分子等を構成する原子一つひとつの運動をニュートン方程式に従い数値的に解いて、分子の運動を追跡する手法 - ※3 AlphaFold2
英国DeepMind社の研究グループが開発した、タンパク質のアミノ酸配列を入力として立体構造を予測するAI
5. 論文情報
6. 研究グループ
- 分子科学研究所
- 岡山大学
7. 研究サポート
本研究は、日本学術振興会・科学研究費補助金(若手研究JP23K14160,基盤研究(B)JP22H02595)、公益財団法人・長瀬科 学技術振興財団の支援を受けて実施しました。また、本研究の分子シミュレーションとAlphaFold2による構造予測の一部には、自然科学研究機構岡崎共通研究施設・計算科学研究センターを用いました(課題番号:22-IMS-C189,23-IMS-C201)。
8. 問い合わせ先
- 岡崎 圭一(おかざき けいいち)
分子科学研究所・計算科学研究センター / 総合研究大学院大学、准教授
電子メール:keokazaki@ims.ac.jp - 山下 敦子(やました あつこ)
岡山大学 学術研究院医歯薬学域(薬)、教授
電子メール:a_yama@okayama-u.ac.jp - 自然科学研究機構・分子科学研究所 研究力強化戦略室 広報担当
電子メール:press@ims.ac.jp - 国立大学法人 岡山大学総務・企画部広報課
電子メール:www-adm@adm.okayama-u.ac.jp - 総合研究大学院大学 総合企画課 広報社会連携係
電子メール:kouhou1@ml.soken.ac.jp
