2020.09.30
縄文人が感染していた古代ウイルスのゲノム配列を特定 ~縄文人ウイルスから解き明かすウイルス進化過程~
プレスリリース
遺伝学コース
Identification of ancient viruses from metagenomic data of the Jomon people
掲載誌: Journal of Human Genetics
DOI: 10.1038/s10038-020-00841-6


概要
過去に日本列島で生活していた縄文人(1)のゲノム配列(2)を調べることにより、縄文人のルーツや目の色、お酒に強いかなど、さまざまなことがわかってきました。一方で、縄文人がどのようなウイルスに感染していたのかをはじめとしてわからないことも種々残されています。
情報・システム研究機構 国立遺伝学研究所の西村瑠佳さん (総研大遺伝学専攻大学院生) と井ノ上逸朗教授らの研究グループは、縄文人の歯髄から得られたDNAを用い、そこに含まれるウイルスのゲノム配列を決定し、解析を行いました。その結果、11種類のウイルスのゲノム配列が見つかりました。中でもSiphovirus contig89 (CT89) (3)と呼ばれるヒトの口腔内に生息するウイルスについては完全長のゲノム配列データを得ることができました。さらに現代のCT89ウイルスとゲノム配列を比較した結果、本研究で見つかったCT89のゲノム配列は、CT89の祖先型ゲノム配列を反映していることが示唆されました。
今後は、CT89ウイルスがどれくらいの速さで進化してきたのか、どのような進化過程を辿ったのかなどを詳細に解析するとともに、糞石をはじめとする縄文人化石の他の部位にも注目し、縄文人に感染していたと思われる古代ウイルスを多く見つけていく予定です。
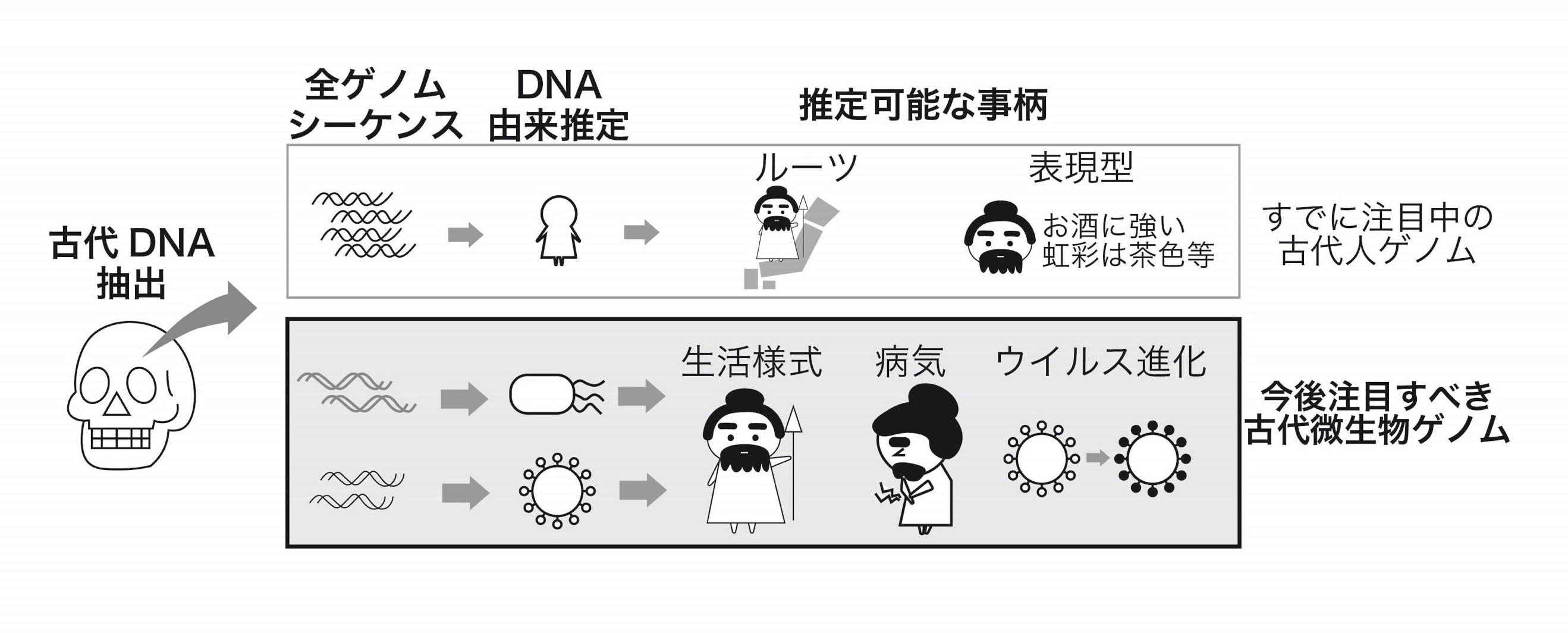
古代人骨の化石に残存するDNAを抽出し、そのゲノム配列を調べることで、どの生物由来のDNAか推定できる。多くのDNAは古代人に由来し、それらの情報によって古代人のルーツや表現型などの解析が盛んに行われてきた。
一方で、古代人骨からDNAを採取すると、古代人に感染していた細菌やウイルスに由来するDNAも含まれていることがわかってきた。細菌やウイルスのゲノム情報に注目することで、古代人の生活様式や病気、ウイルス進化について推定できる。
成果掲載誌
本研究成果は、日本人類遺伝学会誌「Journal of Human Genetics」に2020年9月30日午前10時(日本時間)に掲載されます。
論文タイトル: Identification of ancient viruses from metagenomic data of the Jomon people(縄文人メタゲノムデータにおけるウイルス同定)
著者: Luca Nishimura, Ryota Sugimoto, Jun Inoue, Hirofumi Nakaoka, Hideaki Kanzawa-Kiriyama, Ken-ichi Shinoda, Ituro Inoue(?村瑠佳、杉本?太、井上潤、中岡博史、神澤秀明、篠田謙一、井ノ上逸朗)
研究の詳細
研究の背景
研究の背景
古代人骨の化石にはDNAが残存しています。このDNAを採取すると、古代人由来のDNAだけではなく、古代人に感染していたと考えられる細菌やウイルスのDNAも一緒に採取されてくることがわかっています。このウイルスDNAに注目し、古代から現代にかけて起こった塩基配列の変化を調べることによって、ウイルスがどのような進化を遂げたのかを考察することができます。すなわち、このように古代人骨の化石から採取したウイルスの塩基配列を現代のウイルスの塩基配列と比較することで、現代のウイルスの塩基配列のみに注目した際に生じる時間的なバイアスを排除した進化過程の推定が可能になるのです。しかしながら、日本列島で過去に生活していた縄文人に感染していたウイルスをはじめとする微生物の研究は進んでいませんでした。そこで、本研究チームは、縄文人にどのようなウイルスが感染していて、そのウイルスはどのように進化してきたのかを解き明かすことに挑戦しました。
本研究の成果
縄文人骨の歯髄に含まれる全てのDNAの塩基配列を次世代シークエンサー(4)で決定しました。その中で現代のウイルスと類似の塩基配列を持つゲノム配列を探索したところ、11種類のウイルスのゲノム配列を見つけることができました。中でも約3800年前の北海道船泊遺跡の縄文人骨から見つかったSiphovirus contig89 (CT89) というウイルスはほぼ完全なゲノム配列が復元できました。このCT89はヒト口腔内の細菌に感染するファージ(5)であることがわかっています。38から完全長のファージ配列を再構成した例は世界で初めてです。さらに、現生のCT89のゲノムは部分配列しかデータベース上に登録されていませんでしたが、今回の解析により、未知の領域の配列も明らかにすることができたのです(図2)。
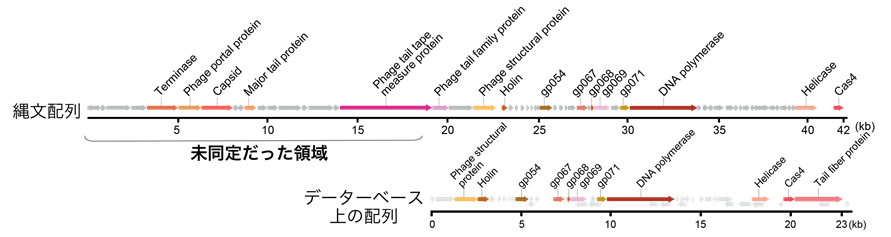
上段に縄文時代のCT89のゲノム構造を、下段にデータベース上のCT89のゲノム構造を示した。矢印はそれぞれのゲノム上に存在する遺伝子を示している。中でも機能を持つと推定されたものは色をつけて示してあり、遺伝子名が記されている。どの遺伝子もウイルスに特徴的な遺伝子であることがわかっている。灰色の矢印は推定タンパク質である。また、左上の領域はデータベース上には登録のない、新たに同定された領域である。
本研究で見出した縄文時代のCT89を含め、古代と現代のCT89の塩基配列を比較し、系統解析(6)を行い進化過程の推定をしたところ、縄文時代のCT89はCT89の祖先型に近い塩基配列を有する可能性が示唆されました(図3)。
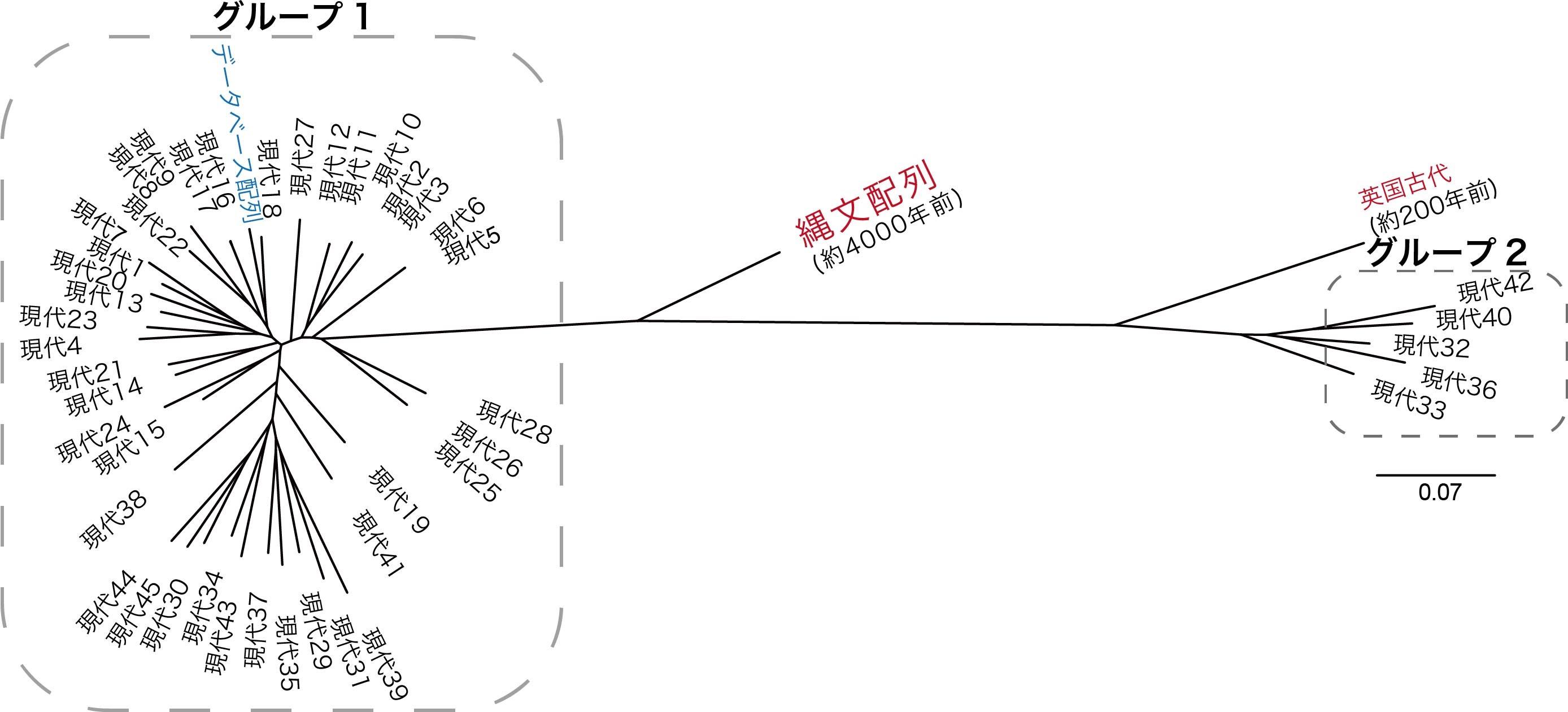
現代の46種類のCT89ゲノム配列と古代の2つのCT89ゲノム配列を用いて系統樹を作成した。(図中の現代1?46は現代の46種類の配列、データベース配列はデータベース上に登録のある配列を示す。)系統樹の各枝の長さが短ければ短いほど、ゲノム配列同士が似通っていることを示す。ここでは、現代の配列では大きく二つのグループに分かれ、古代の配列はそれとは異なっていることがわかる。特に、縄文時代の配列はどちらのグループからも離れていることから、現代の配列と大きく異なり、CT89の祖先のゲノム配列を反映している可能性が高い。
今後の期待
本研究により、縄文人の口腔内に含まれるウイルスを初めて明らかにすることができました。この成果は縄文人をはじめとする古代人に感染していた微生物に関する研究の先駆けとなるものです。今後は、得られたウイルス情報をもとに、ウイルス進化に関する知見を増やすことを目指します。
本成果は、ウイルスの起源を解き明かすことに寄与するだけでなく、感染症を引き起こすウイルス進化の予想につながることが期待されます。
用語解説
(1). 縄文人
約2900~16000年前に日本列島で生活していたとされる古代人。古代人化石の年代は年代測定技術を用いることで推定が可能である。
(2). ゲノム配列
ある生物の持つ全てのDNA塩基配列を指す。
(3). Siphovirus contig89 (CT89)
健康な現代人の口腔内から見つかったウイルスの一種。
(4). 次世代シーケンサー
数億に及ぶDNAの断片を高速に配列決定できる装置の総称であり、この装置を用いることによって大量のゲノム配列を決定することが可能となる。
(5). ファージ
ウイルスの中でも細菌を宿主とするウイルス。
(6). 系統解析
塩基配列やアミノ酸配列を用いて生物の進化過程を推定する解析のこと。生物種間や生物種内での塩基配列やアミノ酸配列の違いを調べることによって、祖先が辿ってきた進化過程を推測することができる。
(7). 系統樹
系統解析の一つで、生物間の類縁関係を塩基配列組成に基づいて樹木状に図示したもの。枝長が短い、つまり塩基配列が似ているものほど近縁であると推定される。
研究体制と支援
本研究は、科研費(18H05506)によって支援されました。本研究で用いた縄文人由来の塩基配列データはヤポネシアゲノムプロジェクトで得られたデータです。
問い合わせ先
<研究に関すること>
国立遺伝学研究所 人類遺伝研究室
<報道担当>
国立遺伝学研究所 リサーチ・アドミニストレーター室 広報チーム
総合研究大学院大学 総合企画課 広報社会連携係
TEL: 046-858-1584 メール: kouhou1@ml.soken.ac.jp
※時節柄、Zoom会議での取材にも対応できますので、Zoom会議をご希望の場合には、その旨お知らせください。
